Čárový kód života
 Již roku 2003 skupina zoologů z Guelphské univerzity v kanadském Ontariu navrhla čárový kód života (DNA Barcoding of Life) jako způsob identifikace organismů na Zemi (obr. 2). To by znamenalo revoluci v taxonomické práci (viz Vesmír 87, 294, 2008/5) a především u „nesnadných“ skupin umožnilo určit organismus pomocí malých, standardizovaných kousků genomu, náhradou za morfologické znaky. Cílová představa je lákavá: Do ruční čtečky (nemůžu vynechat pěkný anglický termín „handheld barcoder“, obr. 1) se vloží kousek zkoumaného organismu a výsledkem je jméno druhu, jeho zařazení, geografické rozšíření a další užitečné informace.
Již roku 2003 skupina zoologů z Guelphské univerzity v kanadském Ontariu navrhla čárový kód života (DNA Barcoding of Life) jako způsob identifikace organismů na Zemi (obr. 2). To by znamenalo revoluci v taxonomické práci (viz Vesmír 87, 294, 2008/5) a především u „nesnadných“ skupin umožnilo určit organismus pomocí malých, standardizovaných kousků genomu, náhradou za morfologické znaky. Cílová představa je lákavá: Do ruční čtečky (nemůžu vynechat pěkný anglický termín „handheld barcoder“, obr. 1) se vloží kousek zkoumaného organismu a výsledkem je jméno druhu, jeho zařazení, geografické rozšíření a další užitečné informace.
Celá věc je však poněkud složitější, neboť vyžaduje minimálně dobře vybavenou molekulární laboratoř a obrovskou databázi s informacemi nejlépe o všech organismech na světě. Celý proces by pak měl být rychlý, levný a dobře dostupný. Původní idea Paula Heberta, který s nápadem čárového kódu života přišel jako první, byla přesně taková. Napadla ho v supermarketu při procházení mezi policemi se zbožím. Uvažoval, zda by se nedala jedinečná informace v písmenech DNA využít podobným způsobem jako kombinace tenčích a silnějších čar na zboží. Zní to jako utopie?
Deset důvodů pro DNA identifikaci
Konsorcium pro čárový kód života (Consorcium for the Barcode of Life, CBOL) stanovilo deset hlavních důvodů, proč se zabývat DNA identifikací. Zde si shrňme jejich hlavní poselství.
 I když se sekvenační technologie v současnosti výrazně zdokonalily, stále není a dlouho ještě nebude možné sekvenovat a složit celý genom eukaryotických organismů. Proto se jako nejlepší řešení ukazuje identifikace druhů na základě krátkých fragmentů; v nejlepším případě by mohl stačit jen jeden. Po zavedení do praxe by čárový kód umožňoval např. rychle identifikovat v potravinách obsažené rostliny či živočichy pocházející z nezákonného obchodu, detekovat komerční produkty vyrobené z ohrožených druhů či přímo při transportech zachytit pašované druhy vzácných a ohrožených organismů. Důležité je, že identifikace založená na molekulárním čárovém kódu je nezávislá na vývojovém stadiu organismu, které máme k dispozici. Jednoduše nezáleží na tom, jestli celníci na letišti zabaví vejce, semena, celý rostlinný či živočišný druh nebo jen jeho část. Čárový kód umožňuje spolehlivě rozlišit druhy, které vypadají velmi podobně a na základě morfologických znaků jsou často zaměňovány. Příkladem mohou být komáři rodu Anopheles. Celkem je známo zhruba 430 druhů, pouze několik málo z nich však přenáší malárii na člověka.
I když se sekvenační technologie v současnosti výrazně zdokonalily, stále není a dlouho ještě nebude možné sekvenovat a složit celý genom eukaryotických organismů. Proto se jako nejlepší řešení ukazuje identifikace druhů na základě krátkých fragmentů; v nejlepším případě by mohl stačit jen jeden. Po zavedení do praxe by čárový kód umožňoval např. rychle identifikovat v potravinách obsažené rostliny či živočichy pocházející z nezákonného obchodu, detekovat komerční produkty vyrobené z ohrožených druhů či přímo při transportech zachytit pašované druhy vzácných a ohrožených organismů. Důležité je, že identifikace založená na molekulárním čárovém kódu je nezávislá na vývojovém stadiu organismu, které máme k dispozici. Jednoduše nezáleží na tom, jestli celníci na letišti zabaví vejce, semena, celý rostlinný či živočišný druh nebo jen jeho část. Čárový kód umožňuje spolehlivě rozlišit druhy, které vypadají velmi podobně a na základě morfologických znaků jsou často zaměňovány. Příkladem mohou být komáři rodu Anopheles. Celkem je známo zhruba 430 druhů, pouze několik málo z nich však přenáší malárii na člověka.
Mohlo by se zdát, že čárový kód bude jen praktickou pomůckou především pro mimovědecké použití. Opak je však pravdou. V současnosti je na Zemi kolem 2 milionů druhů organismů, což je pouze malá část předpokládaného celkového množství, které se odhaduje na 10 milionů (číslo je nutno brát jen velmi orientačně, neboť předpoklady se značně liší podle taxonomického pojetí).
 Výsledným produktem snažení by měla být „encyklopedie života na Zemi“, která by zahrnovala unikátní čárové kódy organismů, odkazy na jejich muzejní položky, jména a zařazení organismů a další důležité údaje. To vše veřejně přístupné on-line v internetové databázi. Knihovna digitálních čárových kódů bude umožňovat jednoznačné srovnání druhů založené na digitální identifikaci, která je mnohem přesnější než dosud používaný analogový postup určování pomocí slov, tvarů a barev. V neposlední řadě je potřeba vyzdvihnout v současnosti poněkud opomíjenou úlohu muzejních sbírek rostlin a živočichů, které sehrávají v identifikaci organismů hlavní úlohu srovnávacího materiálu.
Výsledným produktem snažení by měla být „encyklopedie života na Zemi“, která by zahrnovala unikátní čárové kódy organismů, odkazy na jejich muzejní položky, jména a zařazení organismů a další důležité údaje. To vše veřejně přístupné on-line v internetové databázi. Knihovna digitálních čárových kódů bude umožňovat jednoznačné srovnání druhů založené na digitální identifikaci, která je mnohem přesnější než dosud používaný analogový postup určování pomocí slov, tvarů a barev. V neposlední řadě je potřeba vyzdvihnout v současnosti poněkud opomíjenou úlohu muzejních sbírek rostlin a živočichů, které sehrávají v identifikaci organismů hlavní úlohu srovnávacího materiálu.
Výběr vhodné oblasti genomu
 Co je to vlastně „Barcode of Life“? Podle původní představy krátký, univerzální úsek DNA z určité oblasti genomu sloužící k identifikaci organismu na druhové úrovni. Tento předpoklad byl do určité míry splněn u živočichů, u nichž byl univerzálním markerem stanoven mitochondriální gen cytochrom c oxidázy 1 (COI). Pro jeho použití hovoří zejména počet kopií v buňce. Jaderný genom je v buňce většinou uložen ve dvou kopiích, mitochondriální ve 100 až 10 000 kopiích. To jasně favorizuje mitochondriální genom, neboť i u velmi degradovaných vzorků DNA je naděje na získání sekvenovatelné kopie daného úseku. Dalším důvodem je velká mezidruhová rozmanitost genu COI. I mezi velmi blízce příbuznými druhy se sekvence liší pětinásobně až desetinásobně v porovnání s geny jadernými. Současně je vnitrodruhová variabilita genu COI velmi nízká. Malá vnitrodruhová a velká mezidruhová rozmanitost ukazují na zřetelné genetické hranice mezi druhy a umožňují přesnou identifikaci právě pomocí tohoto úseku.
Co je to vlastně „Barcode of Life“? Podle původní představy krátký, univerzální úsek DNA z určité oblasti genomu sloužící k identifikaci organismu na druhové úrovni. Tento předpoklad byl do určité míry splněn u živočichů, u nichž byl univerzálním markerem stanoven mitochondriální gen cytochrom c oxidázy 1 (COI). Pro jeho použití hovoří zejména počet kopií v buňce. Jaderný genom je v buňce většinou uložen ve dvou kopiích, mitochondriální ve 100 až 10 000 kopiích. To jasně favorizuje mitochondriální genom, neboť i u velmi degradovaných vzorků DNA je naděje na získání sekvenovatelné kopie daného úseku. Dalším důvodem je velká mezidruhová rozmanitost genu COI. I mezi velmi blízce příbuznými druhy se sekvence liší pětinásobně až desetinásobně v porovnání s geny jadernými. Současně je vnitrodruhová variabilita genu COI velmi nízká. Malá vnitrodruhová a velká mezidruhová rozmanitost ukazují na zřetelné genetické hranice mezi druhy a umožňují přesnou identifikaci právě pomocí tohoto úseku.
 Velmi důležitým rysem obecně živočišného mitochondriálního genomu, a tedy i genu COI, je nepřítomnost intronů (živočišná mtDNA občas sice introny obsahuje, ale je to velmi řídký jev, jakási výjimka potvrzující pravidlo), což umožňuje velmi jednoduché namnožení zvoleného úseku. Jedna z prvních studií kanadského zoologa Paula Heberta ukázala, že je pomocí cytochrom c oxidázy 1 možné rozeznat všech 260 ptačích druhů, které byly do studie zařazeny. Mezi další skupiny, kde se COI gen osvědčil jako vhodný diskriminační úsek, patří ryby a motýli. Problematické ovšem zůstávají skupiny s nízkou sekvenční variabilitou (koráli či mořské sasanky), u kterých nelze blízce příbuzné druhy spolehlivě odlišit. Podobně je tomu s druhy vzniklými křížením či s druhy nově povstalými.
Velmi důležitým rysem obecně živočišného mitochondriálního genomu, a tedy i genu COI, je nepřítomnost intronů (živočišná mtDNA občas sice introny obsahuje, ale je to velmi řídký jev, jakási výjimka potvrzující pravidlo), což umožňuje velmi jednoduché namnožení zvoleného úseku. Jedna z prvních studií kanadského zoologa Paula Heberta ukázala, že je pomocí cytochrom c oxidázy 1 možné rozeznat všech 260 ptačích druhů, které byly do studie zařazeny. Mezi další skupiny, kde se COI gen osvědčil jako vhodný diskriminační úsek, patří ryby a motýli. Problematické ovšem zůstávají skupiny s nízkou sekvenční variabilitou (koráli či mořské sasanky), u kterých nelze blízce příbuzné druhy spolehlivě odlišit. Podobně je tomu s druhy vzniklými křížením či s druhy nově povstalými.
Složitější hledání u rostlin
K zoologům se brzy přidali i botanici. Roku 2005 Vincent Savolainen optimisticky uvauvažoval o čárovém kódu DNA jako o levném a rychlém nástroji pro identifikaci všech odhadovaných 10 milionů druhů na Zemi.
Identifikační možnosti daného lokusu čárového kódu se u rostlin ukazují mnohem složitější než u živočichů, protože rostlinné genomy jsou komplexnější a druhové bariéry jsou u rostlin hůře definovatelné. Ani gen COI není možné využít, protože rostlinný mitochondriální genom je příliš konzervativní neboli nedostatečně mezidruhově variabilní. Některé žádoucí vlastnosti srovnatelné s mitochondriálním genomem živočichů však má genom chloroplastů. Chloroplastová DNA se také například v buňce vyskytuje ve více kopiích.
V srpnu roku 2009 byl publikován souhrnný článek konsorcia pro čárový kód života doporučující pro čárový kód suchozemských rostlin kombinaci dvou chloroplastových genů – rbcL (Rubisco čili ribuloso-1,5-bisfosfátkarboxyláza) a matK (maturáza K). Oba se běžně používají ve fylogenetických studiích rostlin (obr. 5).
Celkem bylo úspěšně testováno 7 úseků (geny matK, rbcL, rpoB a rpoC1, nekódující úseky atpF-atpH, trnH-psbA a psbK-psbI; viz obr. 3) pro 400 druhů vyšších rostlin a zjistilo se, že nejvyšší rozlišovací schopnost mají nekódující úseky psbK-psbI a trnH-psbA (68 a 69 % správně určených druhů), dále geny matK (66 %), rbcL (61 %) následované genem rpoC1 (43 %). Pokud se zvolily dva lokusy, vzrostla rozlišovací schopnost na 59–75 %, pro kombinaci tří lokusů až na 65–76 %. Při použití všech sedmi testovaných úseků byla pravděpodobnost správného zařazení druhu 73 %.
Zdálo by se tedy, že je vše jasné. Vhodné úseky pro čárový kód rostlin byly navrženy a nezbývá nic jiného, než získat rostlinnou DNA, tu namnožit a sekvenovat. Vše jednoduše, rychle a levně.
Proč to není tak jednoduché?
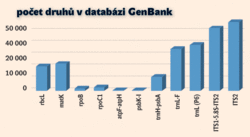
Dvoulokusový čárový kód rbcL/matK se přes svou univerzálnost, vysokou kvalitu získaných sekvencí napříč rostlinnou říší a rozlišovací možnosti ukazuje jako nedostačující. V různých taxonomických skupinách je totiž rozlišovací schopnost vybraných úseků různá. Mnohdy se dosti dramaticky liší jednotlivé geny či hlavně nekódující úseky (obr. 4). Mezi hlavní faktory, které hrají roli při variabilitě úseků, a tudíž úspěšné rozlišitelnosti druhů, patří stupeň hybridizace, polyploidie, typ rozmnožování (problém vzniká u blízce příbuzných agamospermických a autogamických linií), životní historie druhu (dlouhověké organismy mají většinou velmi pomalu mutující DNA) či historie druhu jako takového (rychlá speciace ve velkých populacích vede k udržování ancestrálního polymorfismu).
Tři příklady za všechny
Uveďme si nyní pár příkladů. Jedna z prvních studií čtení čárového kódu DNA u rostlin zahrnovala starobylý řád Cycadales. U něj žádný úsek navržený konsorciem není dostatečně variabilní, aby mohl identifikovat všechny druhy. Jinak je tomu s kombinací několika úseků, ne však jen chloroplastových. Jako ideální se v případě cykasovitých rostlin ukázal jaderný úsek ITS.
 Ve studii rodu krokus, která zahrnovala všech 86 známých druhů, dánští botanici hodnotili 6 úseků chloroplastové DNA. Ukázalo se, že je velmi složité najít vhodné úseky, které by byly dostatečně variabilní. Ani kombinace všech 6 oblastí není schopna spolehlivě identifikovat všechny druhy krokusů, ale pouze 79 %. Hlavním a velmi častým důvodem neúspěchu čtení kódu u rostlin je neshoda mezi vybranými úseky a z toho plynoucí nedostatečné rozlišení (viz např. Vesmír 86, 732, 2007/11). Jeden úsek vypovídá třeba o monofyletické historii dané skupiny a jiný ukazuje na parafylii či polyfylii.
Ve studii rodu krokus, která zahrnovala všech 86 známých druhů, dánští botanici hodnotili 6 úseků chloroplastové DNA. Ukázalo se, že je velmi složité najít vhodné úseky, které by byly dostatečně variabilní. Ani kombinace všech 6 oblastí není schopna spolehlivě identifikovat všechny druhy krokusů, ale pouze 79 %. Hlavním a velmi častým důvodem neúspěchu čtení kódu u rostlin je neshoda mezi vybranými úseky a z toho plynoucí nedostatečné rozlišení (viz např. Vesmír 86, 732, 2007/11). Jeden úsek vypovídá třeba o monofyletické historii dané skupiny a jiný ukazuje na parafylii či polyfylii.
V předchozích dvou příkladech jsme se zabývali použitím čárového kódu DNA u dvou taxonomických skupin; v prvém na úrovni řádu a v druhém na úrovni druhu. Nyní se podívejme na jiný typ studie. V oblasti Střední Ameriky a jižní Afriky, tedy v regionech označovaných jako centra biodiverzity (Vesmír 84, 30, 2005/1), vědci nasbírali přes 1600 rostlin. Získali z nich sekvence 8 různých navržených úseků. Většinu druhů byl překvapivě schopen identifikovat pouze samotný gen matK a například u středoamerických orchidejí se podařilo jen na základě tohoto jediného úseku odhalit kryptické druhy. Tím se matK stal vhodným kandidátem pro identifikaci druhů zapsaných na listině CITES, v praxi tak může posloužit při odhalování pašovaných ohrožených a především kriticky ohrožených druhů.
Quo vadis, barcoding?
DNA barcoding se v současné době v rostlinné říši posunul ze srovnávání vhodnosti různých úseků, především chloroplastového genomu, k hlavním třem praktickým aplikacím. První úlohou čárového kódu na úrovni druhové taxonomie je přispění k identifikaci a především vymezení druhů. Užitečnost tohoto přístupu se projevuje zejména u druhů s velmi jednoduchou morfologií, u velmi drobných rostlin či doposud nedostatečně taxonomicky prozkoumaných a charakterizovaných skupin. Mezi takové patří kupříkladu kryptické druhy mechorostů.
Druhou hlavní aplikací je identifikace neznámých druhů. Ačkoli systematici za posledních 250 let popsali více než 1,7 milionu druhů organismů, je stále více druhů neznámých než známých. Zvyšující se rychlost vymírání poukazuje na potřebu rychlejší identifikace. Čárový kód DNA se ukazuje být vhodnou metodou, která by mohla identifikaci zatím neznámých druhů uspíšit.
Třetím typem studií používajících čárový kód je forenzní botanika, která často potřebuje identifikovat rostlinné zbytky či degradovaný materiál. Specifickým případem je forenzní ekologie, kde DNA barcoding umožňuje určit kořeny rostlin, nejmenší semenáčky či jiná kryptická stadia, jako jsou gametofyty kapraďorostů, ale také rostlinnou potravu býložravců či jednotlivé komponenty léčivých přípravků domorodých kmenů například v Jižní Americe, východní Asii nebo v Africe.
Někteří lidé jsou však k využití čárového kódu DNA stále skeptičtí. Zpočátku převládala představa, že nahradí klasickou taxonomii. Opak je však pravdou. Sekvenování DNA může sice být významným pomocníkem v práci systematického badatele, rozhodně však nenahradí klasické taxonomické přístupy. Ovšem mimo vědeckou oblast se v blízké budoucnosti (po vytipování standardních úseků pro všechny druhy organismů a vytvoření centrální on-line přístupné databáze) může jednat o velmi přínosnou a obecně dobře využitelnou technologii, která se v zatím omezené míře používá již dnes.
SPECIFICKÁ PROBLEMATIKA NĚKTERÝCH KONKRÉTNÍCH ÚSEKŮ DNA
Geny matK, rbcL, rpoB a rpoC1: Chloroplastová DNA obsahuje geny, které jsou pro svou konzervativnost velmi dobře amplifikovatelné i sekvenovatelné. Substituční záměny nukleotidů v DNA jsou u nich vzácné, proto lze snadno porovnat evolučně velice vzdálené druhy. K spolehlivému přiřazení do rodu či druhu však musíme použít vícestupňový proces. Rostlinu nejprve pomocí genů zařadíme do vyšší taxonomické kategorie (řekněme čeledi), poté využijeme některý z nekódujících, a tudíž rychleji mutujících úseků.
Mezigenový mezerník TrnH-psbA: Nekódující úsek trnH-psbA je jedním z nejžhavějších kandidátů rostlinného barcodingu, protože obsahuje velké množství substitučních změn. Práci s ním však ztěžují velmi časté inverzní úseky asociované s palindromickými sekvencemi (obr. 6), které byly nalezeny v různých skupinách krytosemenných rostlin. Proto je nutná velmi pečlivá analýza homologických úseků získaných sekvencí, aby nedošlo k nadhodnocení substitučních událostí především u blízce příbuzných druhů.
Intron TrnL: Intron TrnL je chloroplastový úsek dlouhý 254–767 párů bází, který je hojně využíván pro fylogenetické studie (spolu s intergenovým mezerníkem trnL-F). Je dostatečně variabilní pro taxonomické rozlišení na úrovni čeledí i rodů. Celková rozlišovací schopnost úseku je však poměrně nízká (67,3 %). Přesto je vhodným kandidátem pro barcoding, především v kombinaci s dalšími úseky. Velikou výhodou jsou vysoce konzervované primery a úspěšná amplifikace úseku. Část úseku, smyčka P6 (10–143 bp), je důležitá především pro amplifikaci velmi degradované DNA (např. u velmi starých vzorků). Také srovnávací databáze obsahuje oproti ostatním nekódujícím úsekům velké množství sekvencí trnL, což potenciálnímu uživateli zaručuje, že bude schopen identifikovat svůj vzorek minimálně na rodové úrovni (obr. 5).
Jaderný úsek ITS: Vnitřní transkribovaný mezerník (internal transcribed spacer, ITS) byl stanoven doplňkovým markerem pro skupiny, kde je možno použít přímé sekvenování po provedení amplifikace metodou PCR, tedy bez nutnosti klonování, které by celý postup prodlužovalo a zdražovalo. U některých parazitických rostlin, které mají velmi redukovaný chloroplastový genom, může ITS reprezentovat jediný vhodný marker pro účely barcodingu. Také mnohé recentní studie prokázaly vhodnost použití a rozlišovací schopnosti tohoto úseku, kde rostliny sdílí shodný haplotyp. Problémem úseku ITS však zůstává tzv. spojená evoluce (concerted evolution), která může vést k vytvoření různých paralogních kopií u jednoho druhu. Převažující úseky ITS1-5.8S-ITS2 a ITS2 na obrázku 4 jsou dány právě používáním těchto nukleárních markerů na interspecifické úrovni a studiu spojené evoluce u některých taxonomických skupin. Podobně jako u úseku trnH-psbA je nutno k ITS přistupovat velmi opatrně. Ve většině případů se však odlišné paralogní kopie úseku projeví již dvojitou sekvencí, a také proto připadá jeho použití v úvahu jen u skupin s přímým sekvenováním.
Ke stažení
 článek ve formátu pdf [668,15 kB]
článek ve formátu pdf [668,15 kB]















