Vývoj nových léčiv v počítači
| 3. 9. 2015Počítačová grafika může ovlivnit i tak odlišnou oblast, jakou je farmaceutický průmysl. Díky víceoborové spolupráci na Masarykově univerzitě v Brně vznikl počítačový program CAVER, který je významným pomocníkem právě v oblasti vývoje léčiv a jiných významných chemických látek.
Důležitým stavebním kamenem všech organismů jsou molekuly proteinů. Některé plní stavební funkci, neméně významné jsou však proteinové enzymy, které díky své specifické struktuře katalyzují biochemické reakce probíhající v lidském těle. Je tedy zřejmé, že pro léčbu nemocí a návrat organismu k normálnímu fungování je pomoc proteinů nezbytná.
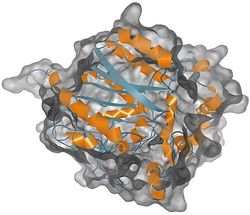
 Protein je složen z řetězce aminokyselin spojených peptidovými vazbami. Řetězec se stáčí do složité prostorové struktury (obr. 2), která je pro každý protein specifická. Rozhodují o ní různé typy slabých interakcí (např. vodíkové vazby nebo van der Waalsovy síly) působících i mezi aminokyselinami, jež spolu v lineární sekvenci řetězce nesousedí.
Protein je složen z řetězce aminokyselin spojených peptidovými vazbami. Řetězec se stáčí do složité prostorové struktury (obr. 2), která je pro každý protein specifická. Rozhodují o ní různé typy slabých interakcí (např. vodíkové vazby nebo van der Waalsovy síly) působících i mezi aminokyselinami, jež spolu v lineární sekvenci řetězce nesousedí.
Proteinová molekula obsahuje takzvaná vazebná místa, která jsou pro katalytické vlastnosti daného proteinu klíčová, protože se k nim vážou molekuly účastnící se katalyzované reakce. Mohou být umístěna na povrchu proteinu, ale často jsou hluboko uvnitř jeho struktury. Nejprve je tedy nutno příslušnou molekulu do vazebného místa dopravit. A právě problémem nalezení vhodných cest se zabývá program CAVER, produkt dlouhodobé spolupráce mezi vědci z Loschmidtových laboratoří Přírodovědecké fakulty a Laboratoře interakce člověka s počítačem Fakulty informatiky na Masarykově univerzitě v Brně.
Program hledá tunely
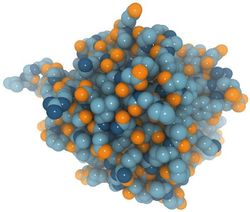
 CAVER je softwarový nástroj zaměřený na analýzu proteinových struktur. Proteiny jsou zkoumány pomocí geometrických metod a algoritmů běžně používaných v oblasti počítačové grafiky. Pokud na protein nahlížíme jako na sekvenci aminokyselin složených z jednotlivých atomů, můžeme přejít ke zjednodušené interpretaci, podle níž se protein skládá ze sady atomů zaujímajících určitou pozici v prostoru. Vstupní datovou sadu pro následnou analýzu si tedy můžeme zjednodušeně představit jako sadu koulí reprezentujících jednotlivé atomy (obr. 3). Takovouto reprezentaci můžeme následně podrobit geometrické analýze, která určí volná místa uvnitř proteinové struktury. V těchto volných místech lze nalézt konkrétní cesty nazývané tunely, vedoucí z vnějšího prostředí proteinu do jeho vazebného místa.
CAVER je softwarový nástroj zaměřený na analýzu proteinových struktur. Proteiny jsou zkoumány pomocí geometrických metod a algoritmů běžně používaných v oblasti počítačové grafiky. Pokud na protein nahlížíme jako na sekvenci aminokyselin složených z jednotlivých atomů, můžeme přejít ke zjednodušené interpretaci, podle níž se protein skládá ze sady atomů zaujímajících určitou pozici v prostoru. Vstupní datovou sadu pro následnou analýzu si tedy můžeme zjednodušeně představit jako sadu koulí reprezentujících jednotlivé atomy (obr. 3). Takovouto reprezentaci můžeme následně podrobit geometrické analýze, která určí volná místa uvnitř proteinové struktury. V těchto volných místech lze nalézt konkrétní cesty nazývané tunely, vedoucí z vnějšího prostředí proteinu do jeho vazebného místa.
 Za první pokus o nalezení tunelů v proteinech lze považovat přístup publikovaný r. 2006 skupinou vědců z Přírodovědecké fakulty Masarykovy univerzity, vedenou profesorem Jiřím Damborským. Jednalo se o první verzi nástroje CAVER. Zvolené řešení však neposkytovalo uspokojivé výsledky, které by byly nezávislé na nastavení parametrů výpočtu. Proto autoři oslovili náš tým z Laboratoře interakce člověka s počítačem s cílem společně překonat limity spojené s první verzí programu. A právě zde se zrodila myšlenka, která se stala základem dalších verzí.
Za první pokus o nalezení tunelů v proteinech lze považovat přístup publikovaný r. 2006 skupinou vědců z Přírodovědecké fakulty Masarykovy univerzity, vedenou profesorem Jiřím Damborským. Jednalo se o první verzi nástroje CAVER. Zvolené řešení však neposkytovalo uspokojivé výsledky, které by byly nezávislé na nastavení parametrů výpočtu. Proto autoři oslovili náš tým z Laboratoře interakce člověka s počítačem s cílem společně překonat limity spojené s první verzí programu. A právě zde se zrodila myšlenka, která se stala základem dalších verzí.
Nově navržená metoda je založena na využití Voroného diagramů, což jsou struktury běžně používané ve výpočetní geometrii (obr. 1). Jméno těmto diagramům dal ukrajinský matematik Georgij















